Spurensuche im Pflanzenmikrobiom mit DNA-Strichcodes
Ein Forscherteam unter der Leitung von Paul Schulze-Lefert hat ein modulares Toolkit entwickelt, um Bakterienstämme zu verfolgen, die Pflanzengewebe im Wettbewerb mit anderen Mitgliedern des Mikrobioms besiedeln. Die Studie wurde jetzt in Nature Microbiology veröffentlicht.

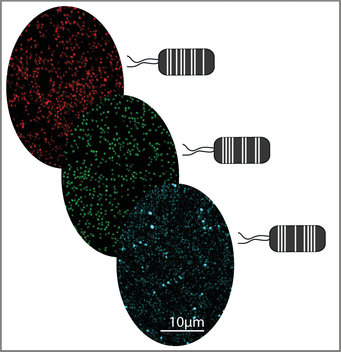
Aus Pflanzen gewonnene Pseudomonas capeferrum-Bakterien, die mit Hilfe des MoBacTag-Toolkits mit verschiedenen fluoreszierenden Tags markiert wurden
Die enorme Vielfalt an Mikroorganismen, die eine gesunde Pflanze in der Natur als Gemeinschaft beherbergt, wird als Pflanzenmikrobiom bezeichnet. Um die Zusammensetzung des mit bloßem Auge unsichtbaren Mikrobioms zu erfassen, wird üblicherweise die DNA-Sequenz eines universellen mikrobiellen Markergens bestimmt, das aus variablen und konservierten Sequenzabschnitten besteht. Auf diese Weise können die mikrobiellen Arten im Mikrobiom anhand der variablen DNA-Sequenzabschnitte voneinander unterschieden werden. Nützliche Aktivitäten von Mitgliedern des Mikrobioms für den Pflanzenwirt, wie z. B. die Mobilisierung von mineralischen Nährstoffen aus dem Boden für die Aufnahme durch die Wurzeln, werden jedoch oft nur von einzelnen Mikrobenstämmen innerhalb einer Art ausgeführt und hängen vom Vorhandensein spezieller Mikrobengenen ab. Die Erstellung von DNA-Sequenz-basierten Mikrobiota-Profilen reicht daher nicht aus, um die tatsächliche genetische Vielfalt der mikrobiellen Gemeinschaft auf dem Pflanzenwirt zu erfassen.
Um dieses Manko zu überwinden, haben Forschende am Max-Planck-Institut für Pflanzenzüchtungsforschung ein modulares Toolkit entwickelt, das als DNA-Strichcode für Bakterienstämme eingesetzt wird. Ein DNA-Strichcode wird zunächst in das Chromosom eines einzelnen Stammes einer Mikrobiomgemeinschaft eingefügt. Bei anschließenden Analysen von Mikrobiomprofilen auf Pflanzen wird der DNA-Strichcode als synthetisches mikrobielles Markergen betrachtet. Neben dem DNA-Strichcode wurden zudem genetische Bausteine für fluoreszierende Proteine eingebaut. Letztere ermöglichen es, mit hochempfindlichen Fluoreszenzdetektoren zu kartieren, wo ein strichcodierter Bakterienstamm in Konkurrenz zu anderen Mikrobiom-Mitgliedern pflanzliches Gewebe besiedelt.

Anschließend führten die Forscher Experimente mit dem pflanzenwachstumsfördernden Bakterium Pseudomonas capeferrum durch, das die Wurzeln der Modellpflanze Arabidopsis besiedelt, sowie mit Varianten des Bakteriums, die sich vom Wildtyp-Stamm nur durch das Fehlen bestimmter Gene unterscheiden. Die entsprechenden Pseudomonas-Gene dämpfen die Immunantworten der Wirtspflanze und verstärken dadurch die Fähigkeit des Bakteriums, Pflanzenwurzeln zu besiedeln, was wiederum deren pflanzenwachstumsfördernde Aktivität erhöht. Die mit DNA-Strichcodes markierten Pseudomonas-Bakterien zeigten zunächst die erwartete unterschiedlichen Fähigkeiten zur Besiedlung von Arabidopsis-Wurzeln, wenn keimfreie Pflanzen mit einzelnen Stämmen beimpft wurden. Überraschend war jedoch das Auftreten qualitativ neuer Eigenschaften der Bakterien, wenn Kombinationen des Wildtyp-Stammes und seiner Varianten zusammen mit einem im Labor zusammengestellten Mikrobiom-Konsortium aus verschiedenen Bakterien auf den Pflanzenwirt geimpft wurden. In der Biologie wird dieses Phänomen auch als emergente Eigenschaft oder Systemeigenschaft bezeichnet.
Die Verwendung von DNA-Strichcodes bestätigte also nicht nur frühere Ergebnisse, sondern deckte auch neue Aktivitäten der bakteriellen Gene auf, die mit herkömmlichen Methoden nicht hätten identifiziert werden können. Das modulare DNA-Strichcode-Toolkit kann nun für die Mikrobiomforschung eingesetzt werden, um den Beitrag einzelner mikrobieller Gene im Kontext mikrobieller Gemeinschaften nicht nur bei Pflanzen, sondern auch in Mikrobiomstudien an Tieren zu untersuchen.
Die Ergebnisse wurden auch in einem „Behind the Paper“ Artikel in Nature vorgestellt.